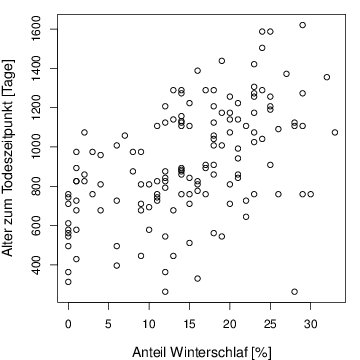
streifen <- read.csv( " hamster.csv " )
n <- nrow(streifen)
streifen.x <- streifen[,1]
streifen.y <- streifen[,2]
l <- 5 # 5 Streifen
mi <- min(streifen.x)
ma <- max(streifen.x)
ra <- ma-mi
# regelmaessige Verteilung im x-Bereich:
xl <- quantile(streifen.x,seq(0,1,length=l+1))
xl[1] <- xl[1]-ra/50
xl[l+1] <- xl[l+1]+ra/50
# Anzahl der Werte in den Abschnitten
nl <- rep(NA,l)
for (i in 1:l){
nl[i] <- sum((streifen.x>=xl[i]) & (streifen.x
pdf("streifen0.pdf",width=5,height=5)
par(mar=c(4,4,1,1))
plot(streifen,xlab="Anteil Winterschlaf [%]",ylab="Alter zum Todeszeitpunkt [Tage]",
cex.lab=1.2)
dev.off()
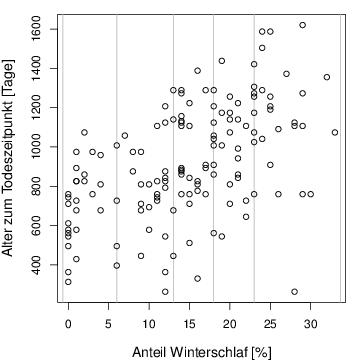
streifen <- read.csv( " hamster.csv " )
n <- nrow(streifen)
streifen.x <- streifen[,1]
streifen.y <- streifen[,2]
l <- 5 # 5 Streifen
mi <- min(streifen.x)
ma <- max(streifen.x)
ra <- ma-mi
# regelmaessige Verteilung im x-Bereich:
xl <- quantile(streifen.x,seq(0,1,length=l+1))
xl[1] <- xl[1]-ra/50
xl[l+1] <- xl[l+1]+ra/50
# Anzahl der Werte in den Abschnitten
nl <- rep(NA,l)
for (i in 1:l){
nl[i] <- sum((streifen.x>=xl[i]) & (streifen.x
pdf("streifen1.pdf",width=5,height=5)
par(mar=c(4,4,1,1))
plot(streifen,xlab="Anteil Winterschlaf [%]",ylab="Alter zum Todeszeitpunkt [Tage]",
cex.lab=1.2,type="n")
abline(v=xl,col=gray(0.7))
points(streifen)
dev.off()
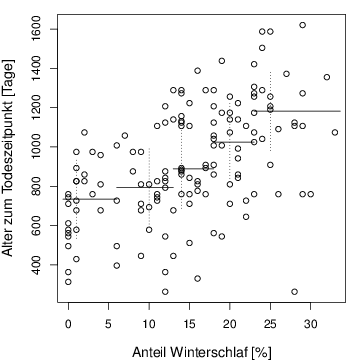
streifen <- read.csv( " hamster.csv " )
n <- nrow(streifen)
streifen.x <- streifen[,1]
streifen.y <- streifen[,2]
l <- 5 # 5 Streifen
mi <- min(streifen.x)
ma <- max(streifen.x)
ra <- ma-mi
# regelmaessige Verteilung im x-Bereich:
xl <- quantile(streifen.x,seq(0,1,length=l+1))
xl[1] <- xl[1]-ra/50
xl[l+1] <- xl[l+1]+ra/50
# Anzahl der Werte in den Abschnitten
nl <- rep(NA,l)
for (i in 1:l){
nl[i] <- sum((streifen.x>=xl[i]) & (streifen.x
streifen.xs <- sort(streifen.x)
streifen.ys <- sort(streifen.y)
nlcum <- c(1,cumsum(nl))
medx <- rep(NA,l) # Mediane in x-Richtung von den Streifen
medy <- rep(NA,l) # Mediane in y-Richtung von den Streifen
for (i in 1:l){
medx[i] <- median(streifen.xs[nlcum[i]:nlcum[i+1]])
medy[i] <- median(streifen.y[(streifen.x>=xl[i]) & (streifen.x
pdf("streifen2.pdf",width=5,height=5)
par(mar=c(4,4,1,1))
plot(streifen,xlab="Anteil Winterschlaf [%]",ylab="Alter zum Todeszeitpunkt [Tage]",
cex.lab=1.2,type="n")
lx <- 200
for (i in 1:l){
segments(xl[i],medy[i],xl[i+1],medy[i])
segments(medx[i],medy[i]-lx,medx[i],medy[i]+lx,lty=3)
}
points(streifen)
dev.off()
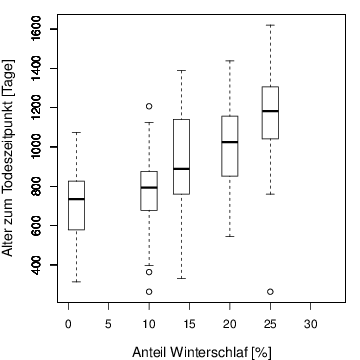
streifen <- read.csv( " hamster.csv " )
n <- nrow(streifen)
streifen.x <- streifen[,1]
streifen.y <- streifen[,2]
l <- 5 # 5 Streifen
mi <- min(streifen.x)
ma <- max(streifen.x)
ra <- ma-mi
# regelmaessige Verteilung im x-Bereich:
xl <- quantile(streifen.x,seq(0,1,length=l+1))
xl[1] <- xl[1]-ra/50
xl[l+1] <- xl[l+1]+ra/50
# Anzahl der Werte in den Abschnitten
nl <- rep(NA,l)
for (i in 1:l){
nl[i] <- sum((streifen.x>=xl[i]) & (streifen.x
streifen.xs <- sort(streifen.x)
streifen.ys <- sort(streifen.y)
nlcum <- c(1,cumsum(nl))
medx <- rep(NA,l) # Mediane in x-Richtung von den Streifen
medy <- rep(NA,l) # Mediane in y-Richtung von den Streifen
for (i in 1:l){
medx[i] <- median(streifen.xs[nlcum[i]:nlcum[i+1]])
medy[i] <- median(streifen.y[(streifen.x>=xl[i]) & (streifen.x
pdf("streifen3.pdf",width=5,height=5)
par(mar=c(4,4,1,1))
plot(streifen,xlab="Anteil Winterschlaf [%]",ylab="Alter zum Todeszeitpunkt [Tage]",
cex.lab=1.2,type="n")
for (i in 1:l){
dat <- streifen.y[(streifen.x>=xl[i]) & (streifen.x
}
dev.off()